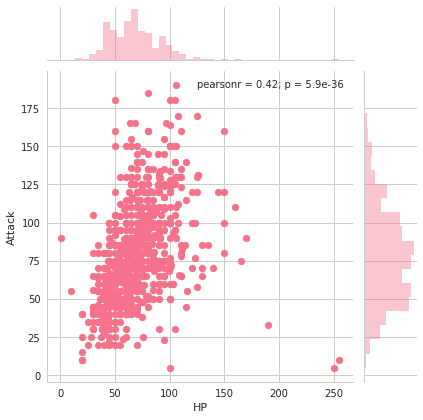
seabornによる統計データ可視化(ポケモン種族値を例に)(2)
plt.figure(figsize=(9, 9))
d = ax.set(xlabel='Physical-Attacker Status', ylabel='Sp-Attacker Status') # d is just for supressing output
plt.figure(figsize=(9, 9))
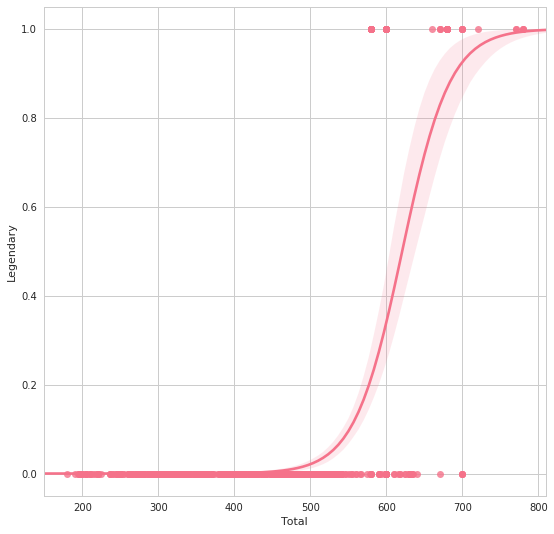
plt.figure(figsize=(9, 9))
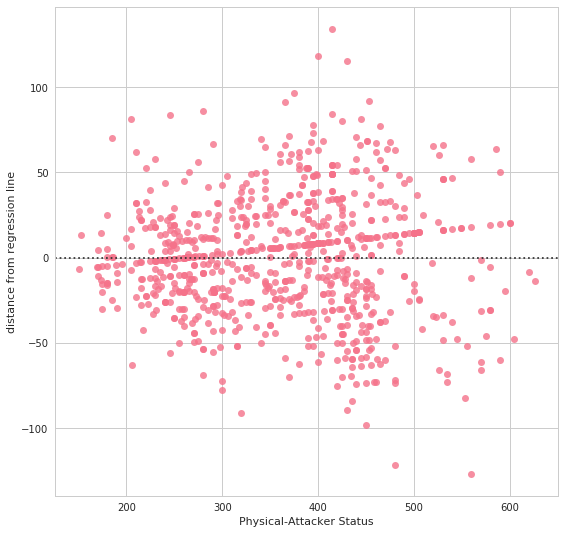
d = ax.set(xlabel='Physical-Attacker Status', ylabel="distance from regression line") # d is just for supressing output
s2 = pkmn["Type 2"]
labels2, levels2 = pd.factorize(s2)
labels1 = levels2.get_indexer(pkmn["Type 1"])
for i in range(len(labels1)):
if(labels2[i]<0): # assign "N/A" as 18th in table
labels2[i] = 18
s[labels1[i] ,labels2[i]] += 1
plt.figure(figsize=(11,9))
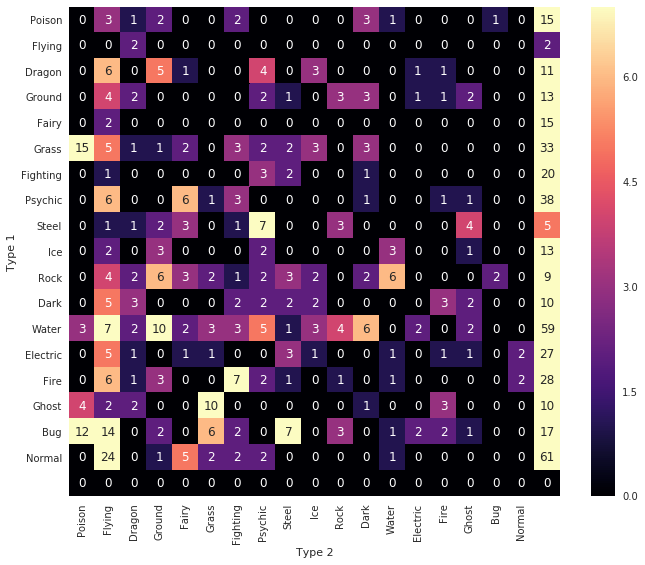
ax = sns.heatmap( s, vmin=0, vmax=7, annot=True, cmap="magma", xticklabels=levels2, yticklabels=levels2)
d = ax.set(xlabel='Type 2', ylabel='Type 1') # d is just for supressing output
import random
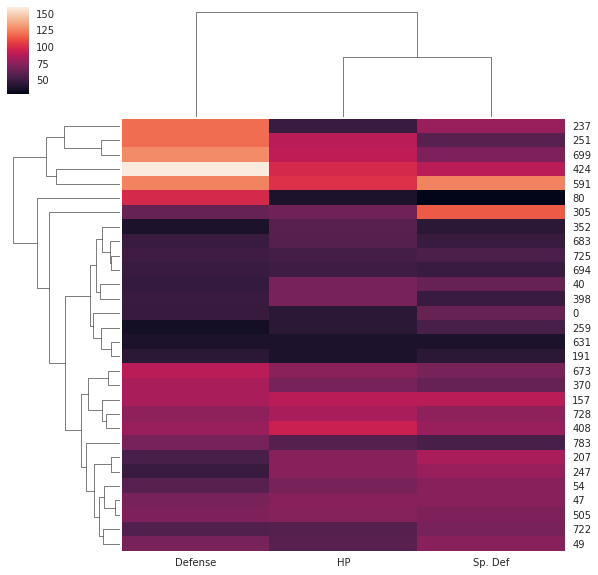
st = pkmn.loc[:, ['HP', 'Defense', 'Sp. Def']].sample(30, random_state=179)
plt.figure(figsize=(11,9))
v = ["HP","Attack","Sp. Atk","Defense", "Sp. Def", "Speed"]
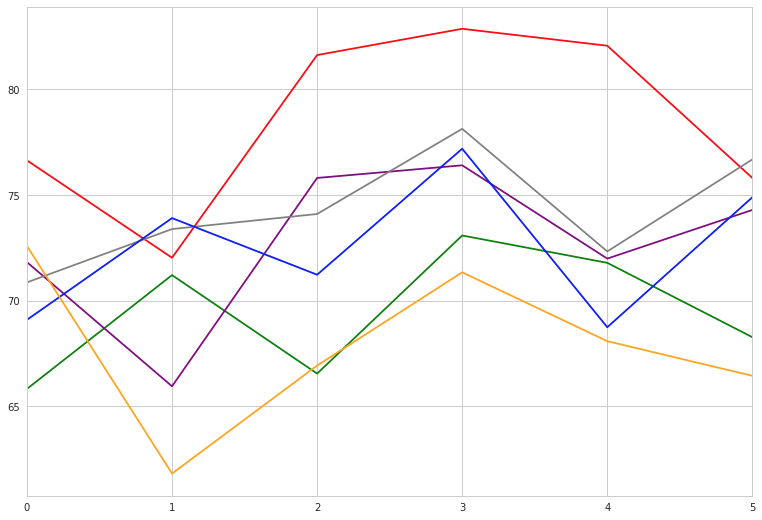
mean = pkmn.groupby(["Generation"])["HP","Attack","Sp. Atk","Defense", "Sp. Def", "Speed"].mean()
#std = pkmn.groupby(["Generation"])["HP","Attack","Sp. Atk","Defense", "Sp. Def", "Speed"].std()
plt.figure(figsize=(13,9))
plt.figure(figsize=(15, 8))
plt.figure(figsize=(16,16))
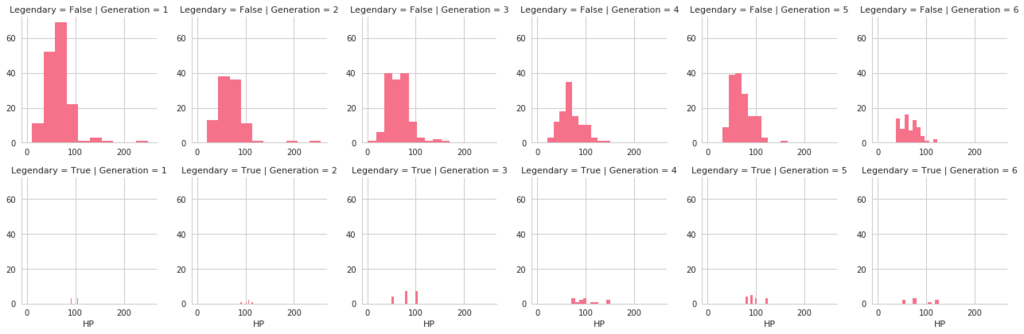 FacetGridは、指定した要素ごとにプロット領域を定義するクラスです。これ単体ではプロットは行われません。 (matplotlibに馴染みのある人であればsubplotをまとめて定義してくれる、と表現すればわかりやすいかもしれません) FacetGridは後述のfactorplot, lmplotで継承されて利用されているようです。
FacetGridは、指定した要素ごとにプロット領域を定義するクラスです。これ単体ではプロットは行われません。 (matplotlibに馴染みのある人であればsubplotをまとめて定義してくれる、と表現すればわかりやすいかもしれません) FacetGridは後述のfactorplot, lmplotで継承されて利用されているようです。In [32]:
plt.figure(figsize=(16,16))
g = g.map(plt.hist, "HP")
plt.figure(figsize=(16,16))
plt.figure(figsize=(16,16))
g = sns.factorplot(x="Generation", y="Sp. Atk", hue="Legendary", data=pkmn, kind="lv", col="Type 1", col_wrap=3
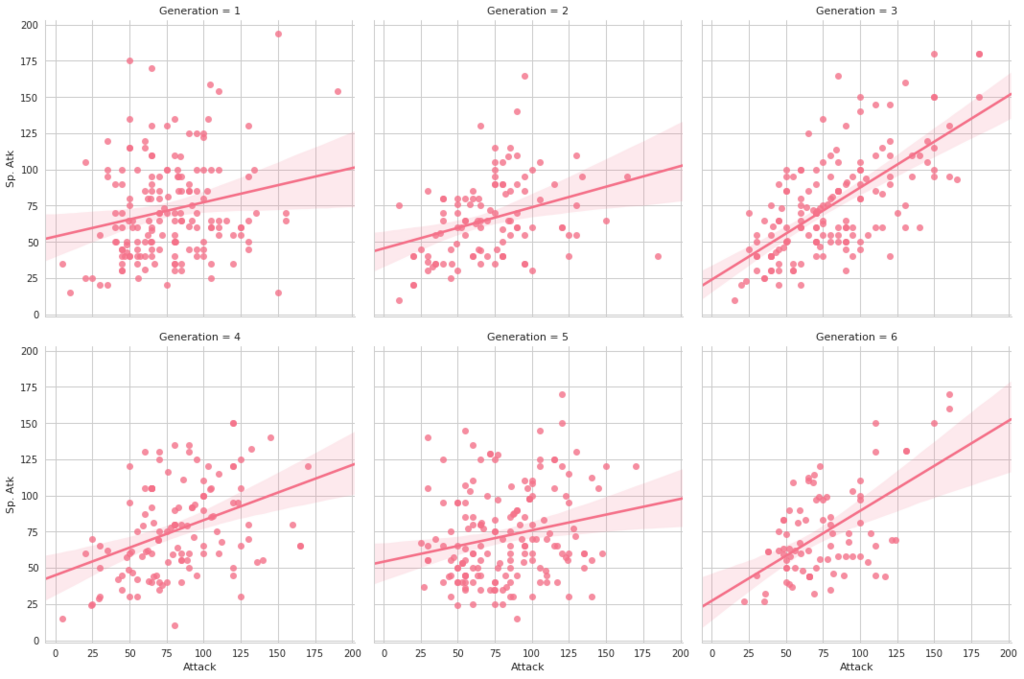
plt.figure(figsize=(16,16))
plt.figure(figsize=(16,16))
#d = pkmn.loc[pkmn.columns.isin(["HP", "Attack", "Sp. Atk", "Defense", "Sp. Def", "Speed"])]
d = pkmn[["HP", "Attack", "Sp. Atk", "Defense", "Sp. Def", "Speed"]]
plt.figure(figsize=(16,16))
plt.figure(figsize=(16,16))
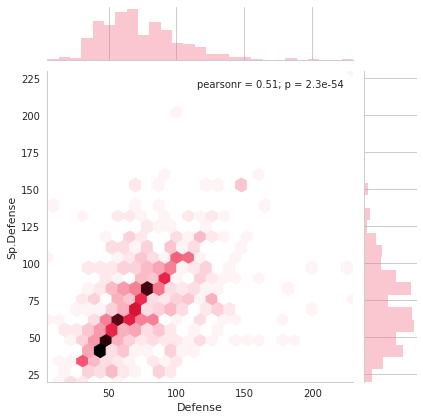
g = sns.jointplot(x = pkmn["Defense"], y = pkmn["Sp. Def"], kind="hex") .set_axis_labels("Defense", "Sp.Defense")
*1:19,19