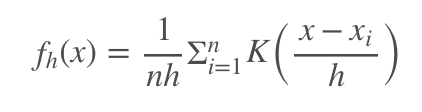Apache2.2から2.4へのhttpd.confの記載方法変更
スタイル変換系論文サーベイ(1)
画像変換、特にStyle変換の論文を読んでまとめていく。随時追記。
A Neural Algorithm of Artistic Style
URL
https://arxiv.org/abs/1508.06576
発表月
2015/8
どんなもの?
・NNをスタイル転写に用いることができることを初めて示した論文。

先行研究に比べて良い点
類似の専攻研究への参照なし
技術・手法のキモ
・学習済みVGG19のパラメータを固定、スタイル画像とコンテンツ入力画像を入力とし、下記の損失関数を最小化するようにホワイトノイズを入力Backpropにより修正していく
モデルと損失
・NNの構成 VGG19をコンテンツ画像、スタイル画像、生成画像に対して利用
・損失の構成
コンテンツ損失: コンテンツ画像と生成画像のあるl層での特徴量の二乗和

スタイル損失: 2つの行列の相関関係を表すグラム行列
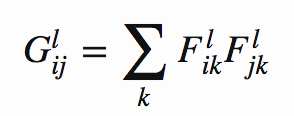
を考え、スタイル画像と生成画像それぞれのグラム行列の二乗平均誤差を

(Gはスタイル画像のグラム行列、Aは生成画像のグラム行列) とする。この E_l の各層での重み付き和をスタイル誤差とする
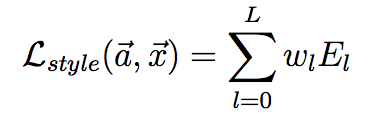
これら2つの誤差の重み付き和
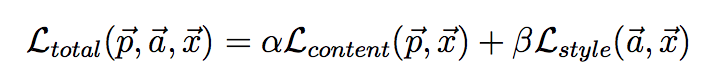
を最小化の対象とする。
有効性検証の方法
- ( 定量的な方法ではなく、様々な画像のスタイル変換を示すことにより)
議論/留意点
・スタイル適用済み画像を生成するためにNNの訓練と同様にBackpropを要するため、非常に時間がかかる
・最適化手法、重み付け方等で様々な改善が後に提案されている
不明点・分からなかったこと
特になし
Perceptual Losses for Real-Time Style Transfer and Super-Resolution
URL
https://arxiv.org/abs/1603.08155
発表月
2016/3
どんなもの?
・A Neural Algorithm of Artistic Styleが提案したスタイル変換をfeed forwardネットワークによりリアルタイムで実施する方法を提案
・超解像度等の画像変換タスクでも従来の"per-pixel"損失を設定した研究に比べ感覚的によりよい結果を得られた
先行研究に比べて良い点
・Semantic SegmentationやDepth Mappingなどの画像変換タスクではピクセルごとの損失(Per Pixel Loss)やCRF Lossを用いていているが、groud-truthとアウトプットの間の「感覚的な(perceptual)」違いを捉えることはできていなかった。
・Gatysらは知覚に沿った損失(Perceptual Loss)を設定し直感的に優れたスタイル変換を達成したが、Backpropにより画像を変換するため生成に時間がかかる
=> 本研究では、両者の良いところを兼ね備えた
- (Per-PixelやCRFでは不可能な) 感覚的な違いを捉えることができ
- feed-forwardにより素早い変換が可能な
手法を提案している
技術・手法のキモ
・画像クラス識別用に事前訓練されたCNNが感覚的/意味的な情報を抽出できることを活かして、損失関数の計算に用いる。(ImageNet訓練済みVGG16)(この点は先行研究と同様)
・直接styleやcontentをpixel単位で比較するのではなく、損失計算用NNのそれぞれの層の出力を比較して損失を計算し、Feed ForwardなNNを訓練する
モデルと損失
モデルの全体構成
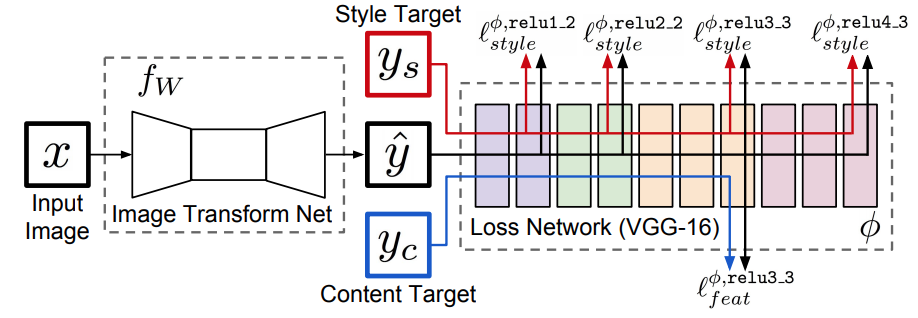
上記NNで、 x: 入力画像 y_s: スタイル画像 y^: xをƒwによって変換した画像 y_c: コンテンツ画像 とする。(スタイル変換では、xとy_cに変換したい画像を、y_sにスタイル対象の画像を入力する)
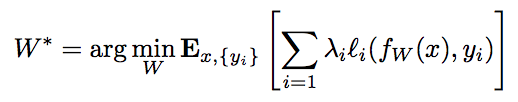
そして、各層で計算された損失の期待値を最小化するƒの重みWを訓練によって獲得する。
- ƒwの構成: Deep Residual Learning for Image RecognitionでResidual Deep CNNを5層重ねて構成する。
- Encoderとして残差付きConvolutionを用いる
- Decoderとして残差付きfractionally strided convolutions(=Deconv)を用いる
- pooling層は用いない
- 非線形関数にはReLUを用いる。
- ただし出力層にはtanhを用いる(0~255の範囲に出力を限定するため)
損失
特徴再現損失:
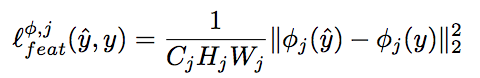
(ここではyはy_c)
スタイル再現損失: グラム行列

に対して、

をスタイル再現損失とする。(ここではyはy_s)
これらの重み付き和を最小化する。
上記の他にも、論文内で超解像度への適用も行っているが、ここでは省略。
有効性検証の方法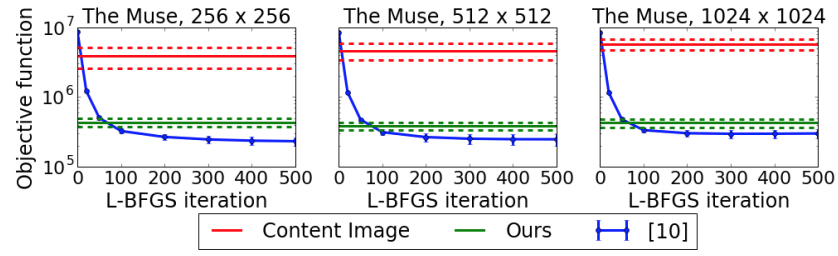 ・定量的にはGatysらの手法に対して、目的関数の差がほぼ同じオーダーに収まることを示した
・定量的にはGatysらの手法に対して、目的関数の差がほぼ同じオーダーに収まることを示した
・また、変換速度についてはGatysらの手法に比べて1000倍近く早いことを示している。 ・定性的には、MS COCOの各種画像をstyle変換した結果を示している。
議論/留意点
・一つのスタイルに対して一つのƒwの訓練を要する ・スタイル変換以外にも超解像度にも適用可能(着彩やsemantic segmentationはfuture workとしている) ・別のデータセットやタスクで訓練された損失計算NNを用いることで、また異なった意味もった生成を行える可能性もある
不明点・分からなかったこと
次に読むべき論文
Let there be Color!: Joint End-to-end Learning of Global and Local Image Priorsfor Automatic Image Colorization with Simultaneous Classification
URL
http://www.f.waseda.jp/hfs/colorization_sig2016.pdf
発表月
2016/7
どんなもの?
- 白黒の写真画像を自動的に着彩する
- 融合層("fusion layer")を導入したことにより、小さな画像に依存する局所的な情報と画像全体から算出される大域的な事前分布をうまく同時に考慮し、既存手法より自然な着彩が可能
- どのような解像度の画像でも着彩可能
先行研究に比べて良い点
- 全体的(global)な情報と部分的(local)な情報を両方考慮したEnd-to-Endな自動着彩(人手による指定一切不要)
- どんな大きさ(解像度)の画像でも処理できる / ラベルを学習に用いることが出来る
技術・手法のキモ
- Globalな特徴を抽出するネットワーク、Localな特徴を抽出するネットワークで別々に画像から特徴を抽出し、それらの結果を融合させるFusion Layer
- Pooling層、FC層を使わないFully Convolutionalな構成(global feature network以外)
- global featureとラベル識別を同時に並行して訓練する訓練手法
モデルと損失


モデル:
- Low Level Feature Network(紫)
Convolution層のみからなる。Global Featureの抽出の入力としても同じ重みのNWを利用する。
f:id:mizti:20180121210600p:plain、以下同じ)
- Global Feature Network(茶色)
ConvとFCからなる。FCを含むため、入力サイズを確定させておく必要があり、このためGlobalへの入力を生成するLow-Levelへの入力画像は224x224にリサイズしておく。
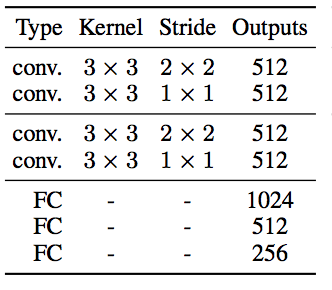
- Mid-Level Feature Network(青色)

Convのみからなる。
- Fusion Layer(赤点線)
Global層の出力と、Mid層の出力を結合したものを入力として1層のFCを適用
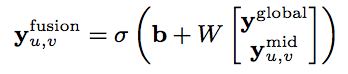 <= 重要
<= 重要
- Colorization Netowrk(緑色)
Deconvolutionを使わず、ConvolutionとUpsampling(Nearest Neighbor)のみからなるデコーダ。
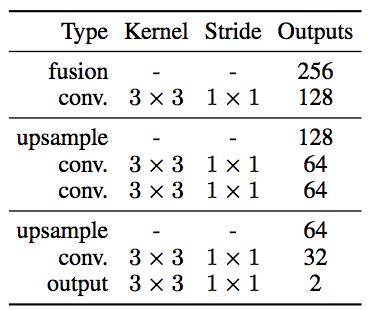
画像はLabカラー形式で扱っているため、L(明度≒モノクロ)を入力とし、出力はaとb*となる。
Batch Normalizationは訓練時のみ適用、訓練終了後は重みに繰り込み
活性化関数はReLU, 最終層はSigmoid
損失:
画像の着彩誤差と同時にラベル識別誤差を足す。

y_colorは二乗和平均(MSE)、y_classはクロスエントロピー (双方が同じ程度の影響になるように、α=1/300程度で設定) (|| ||はフロベニウスノルムの意)
訓練:
最適化アルゴリズムはADADELTAを採用。233万枚の訓練画像をバッチサイズ128で20万イテレーション(11epoch)。256x256にリサイズした画像から毎回224x224でクロップ、50%で左右反転。
有効性検証の方法
・SotAな手法(Cheng2015)、Global Feature無しの着彩、提案手法の3点で着彩を行い、ユーザによる「自然に見える」割合を調査
議論/留意点
- Global Featureのみを別の画像を入力とすることで、全体的なスタイルの転移が可能 (ただし、Semanticにある程度共通点のある画像であることが必要)
- RGB、Lab、YUVの各表色系を比較し、Labが一番良さそうということで採用
- 訓練データにまったく含まれないようなタイプの画像(人の描いたものなど)は上手く着彩できない
不明点・分からなかったこと
- 「Batch Normalizationは訓練時のみ適用、訓練終了後は重みに繰り込み」の具体的な方法
次に読むべき論文
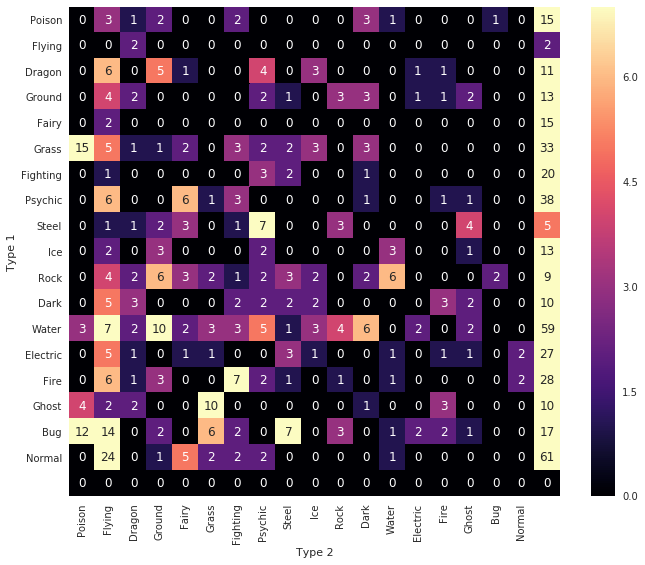
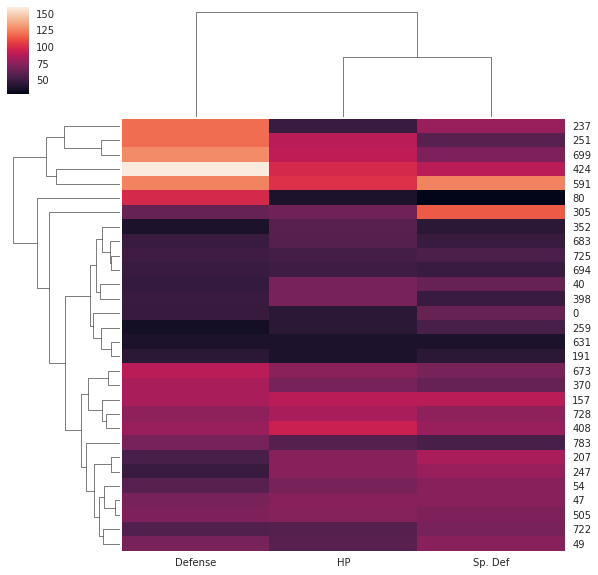
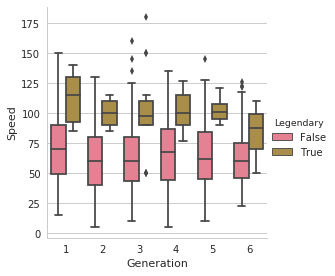
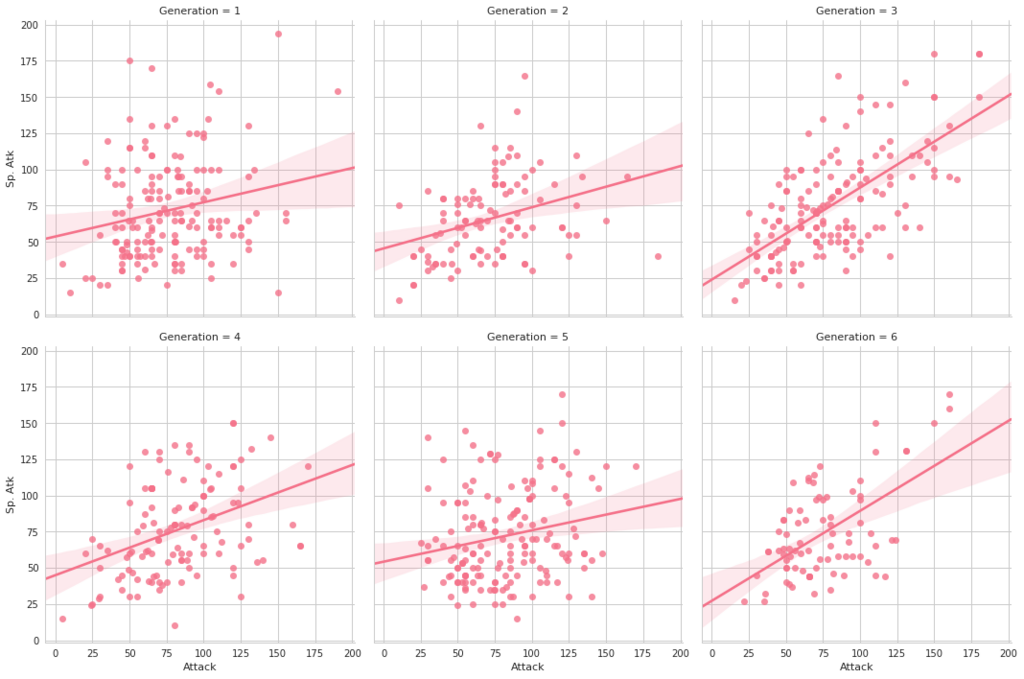
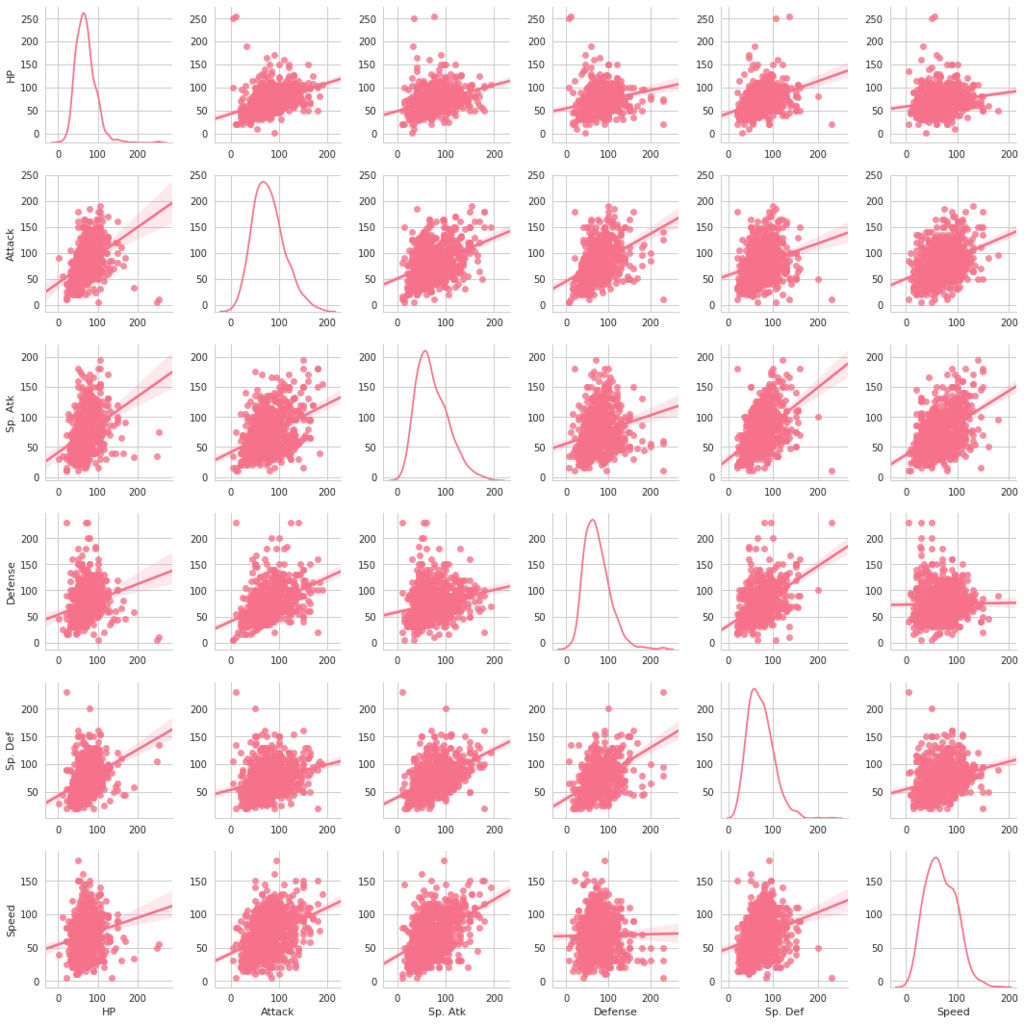
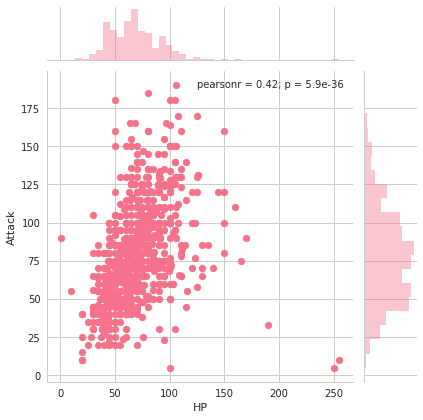
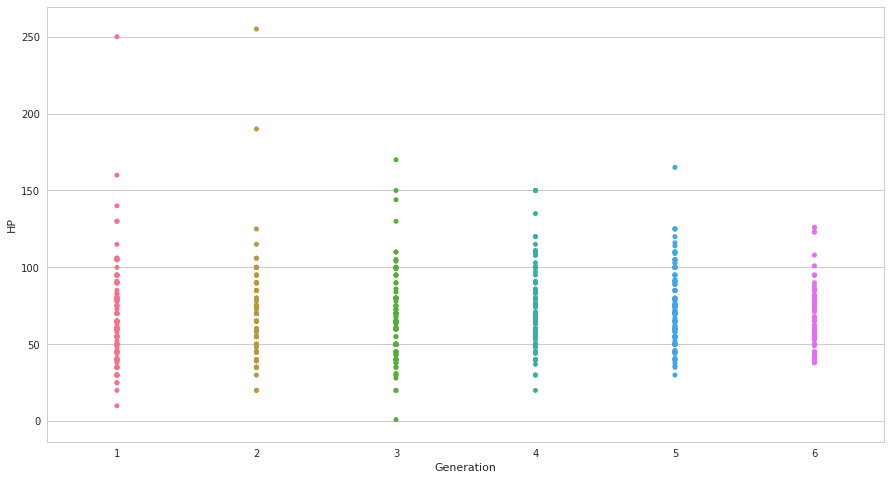
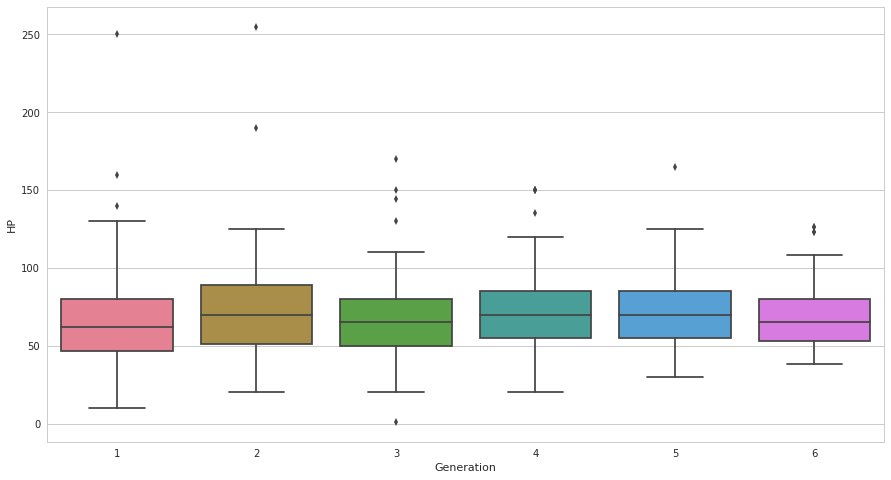
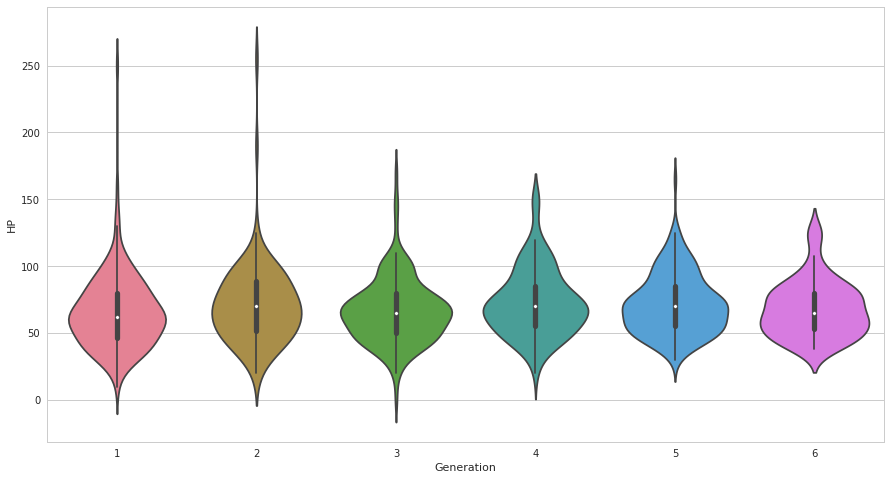
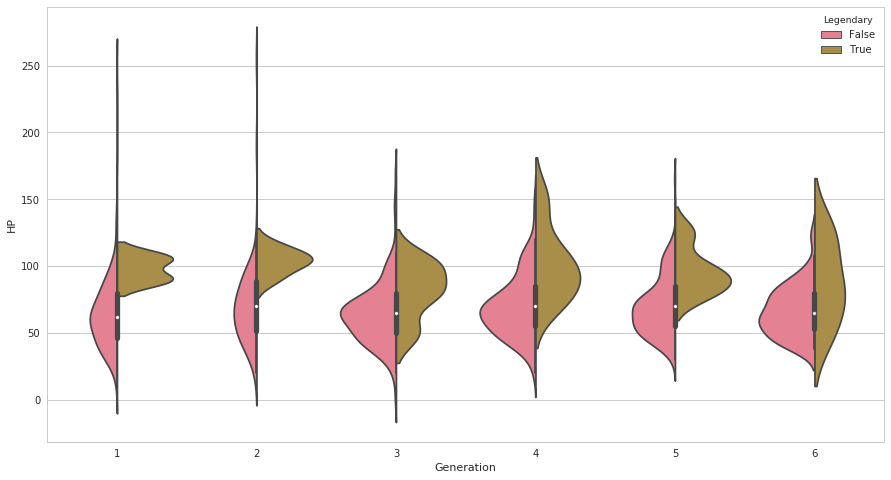
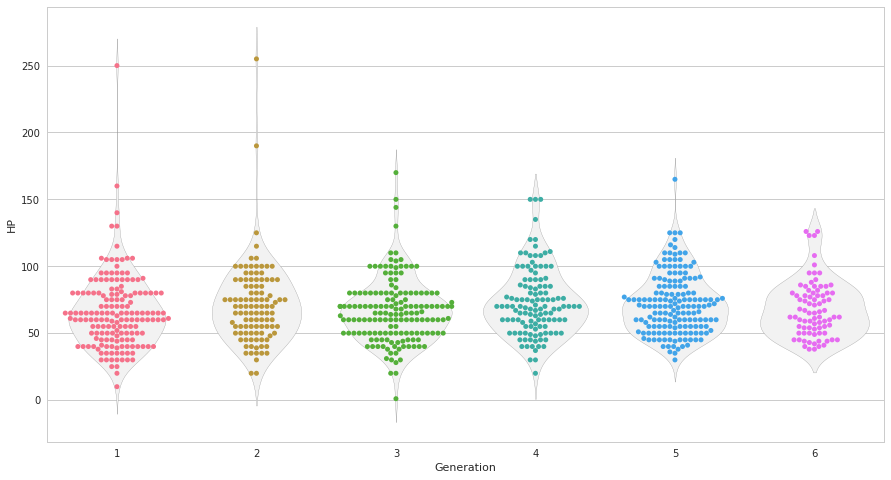
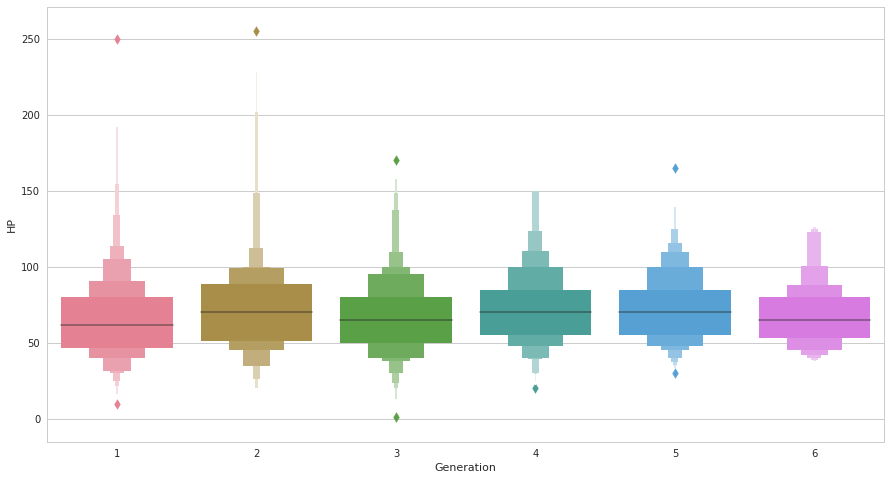
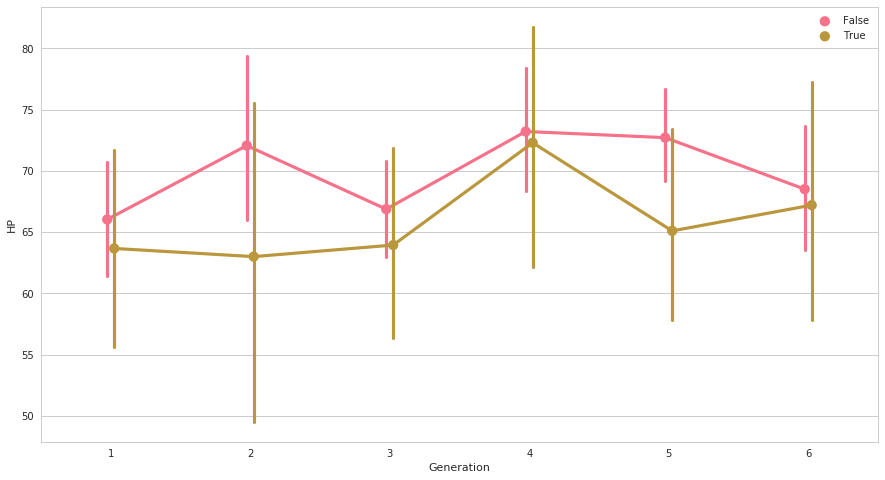
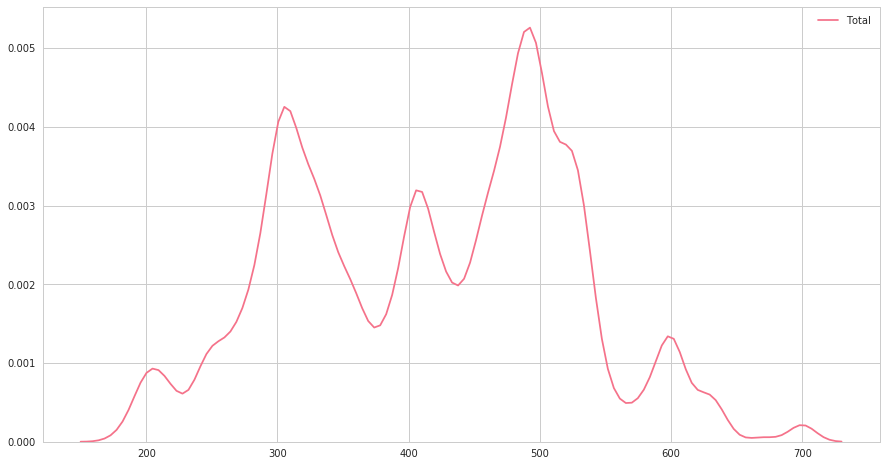
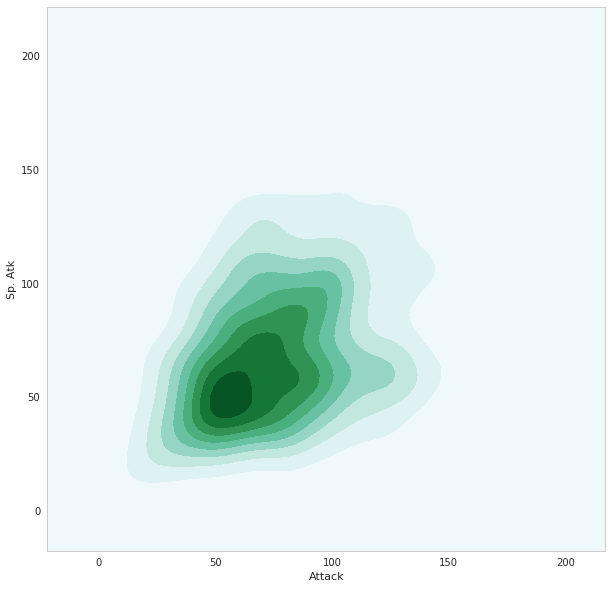
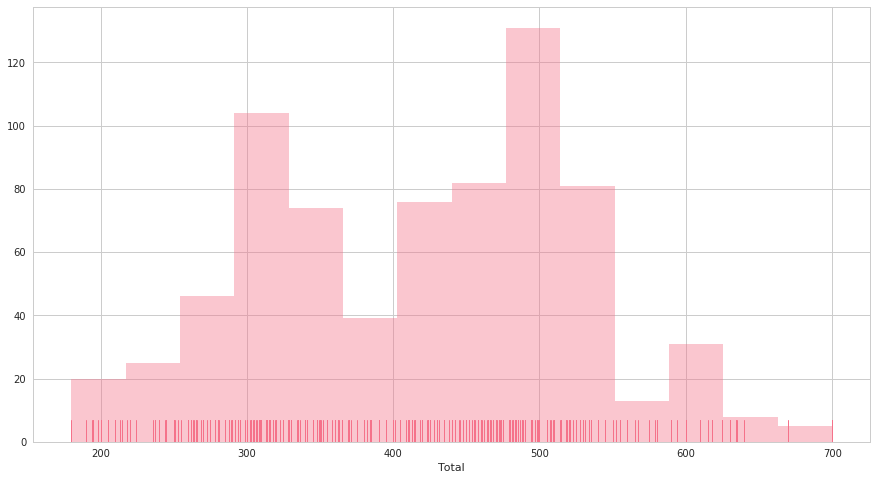
seabornによる統計データ可視化(ポケモン種族値を例に)(2)
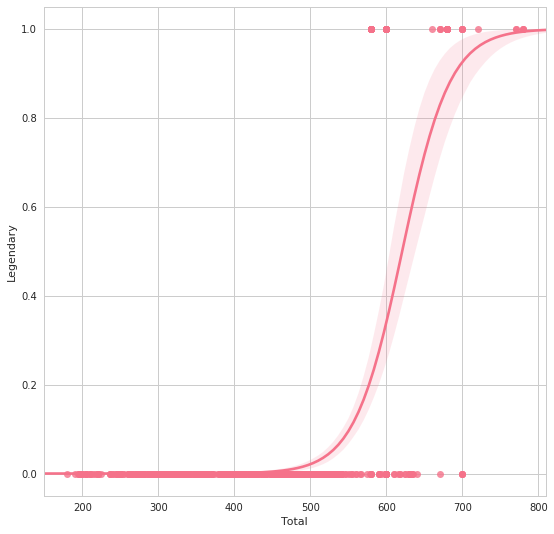
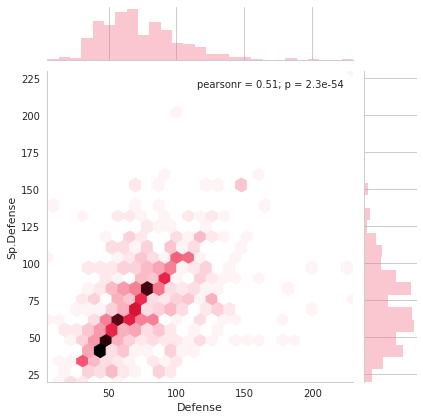
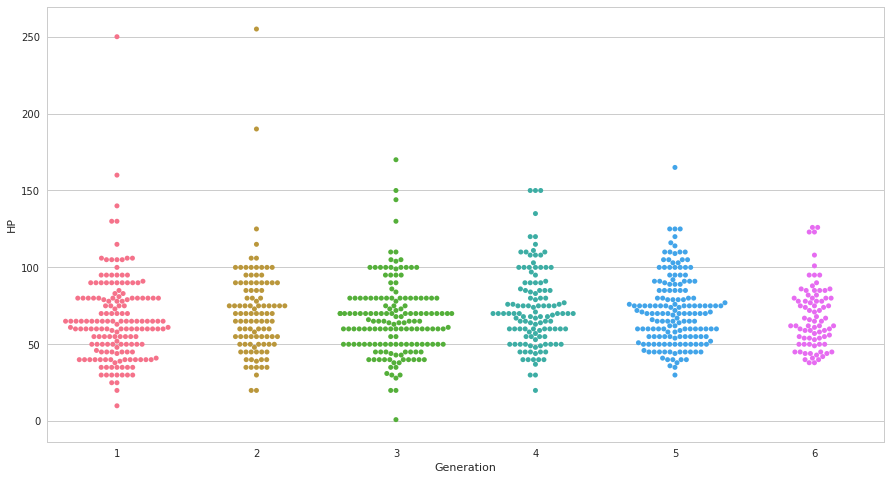
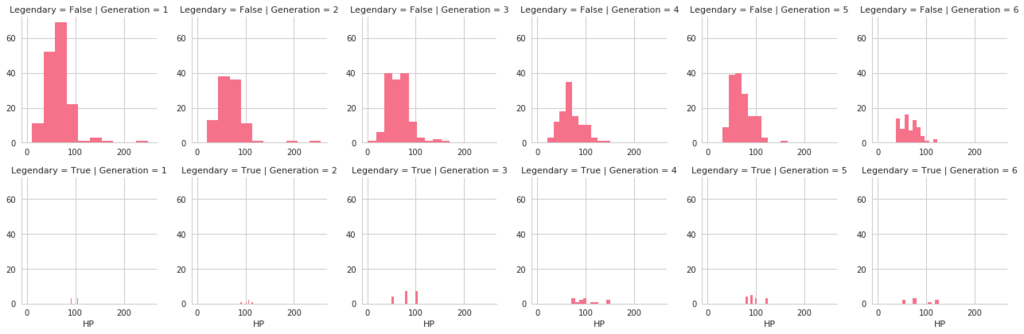 FacetGridは、指定した要素ごとにプロット領域を定義するクラスです。これ単体ではプロットは行われません。 (matplotlibに馴染みのある人であればsubplotをまとめて定義してくれる、と表現すればわかりやすいかもしれません) FacetGridは後述のfactorplot, lmplotで継承されて利用されているようです。
FacetGridは、指定した要素ごとにプロット領域を定義するクラスです。これ単体ではプロットは行われません。 (matplotlibに馴染みのある人であればsubplotをまとめて定義してくれる、と表現すればわかりやすいかもしれません) FacetGridは後述のfactorplot, lmplotで継承されて利用されているようです。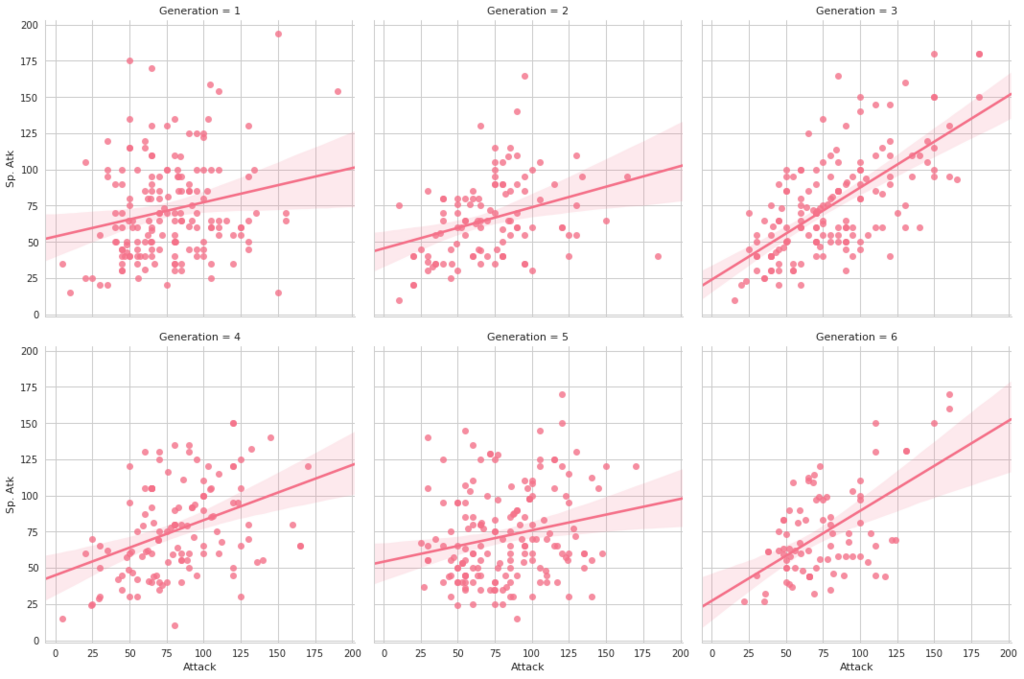
*1:19,19
seabornによる統計データ可視化(ポケモン種族値を例に)(1)
|
#
|
Name
|
Type 1
|
Type 2
|
Total
|
HP
|
Attack
|
Defense
|
Sp. Atk
|
Sp. Def
|
Speed
|
Generation
|
Legendary
|
|
|
0
|
1
|
Bulbasaur
|
Grass
|
Poison
|
318
|
45
|
49
|
49
|
65
|
65
|
45
|
1
|
False
|
|
1
|
2
|
Ivysaur
|
Grass
|
Poison
|
405
|
60
|
62
|
63
|
80
|
80
|
60
|
1
|
False
|
|
2
|
3
|
Venusaur
|
Grass
|
Poison
|
525
|
80
|
82
|
83
|
100
|
100
|
80
|
1
|
False
|
|
3
|
3
|
VenusaurMega Venusaur
|
Grass
|
Poison
|
625
|
80
|
100
|
123
|
122
|
120
|
80
|
1
|
False
|
|
4
|
4
|
Charmander
|
Fire
|
NaN
|
309
|
39
|
52
|
43
|
60
|
50
|
65
|
1
|
False
|
|
#
|
Name
|
Type 1
|
Type 2
|
Total
|
HP
|
Attack
|
Defense
|
Sp. Atk
|
Sp. Def
|
Speed
|
Generation
|
Legendary
|
|
|
121
|
113
|
Chansey
|
Normal
|
NaN
|
450
|
250
|
5
|
5
|
35
|
105
|
50
|
1
|
False
|
|
261
|
242
|
Blissey
|
Normal
|
NaN
|
540
|
255
|
10
|
10
|
75
|
135
|
55
|
2
|
False
|
chainer: トレーニングモジュールの拡張方法まとめ
chainerのチュートリアルをこなしたあと、
- mnistなどライブラリが標準で用意してる以外の独自データセットをどうやって作ったら良いんだろう?
- 複数のモデルが絡むネットワークを同時並行に訓練/評価するにはどうすれば?
といった点で困ることが多くありました。
結果的にはChainerのトレーニング関係のコンポーネントを拡張/自作するのが良さそうなのですが、 それぞれにちょっとずつポイントが有ります。ここでは、トレーニング関連のオブジェクトを概観しながら 今までに書いてきたエントリをまとめます。(今後も増えるかも..)
chainerのトレーニング関連コンポーネント概観
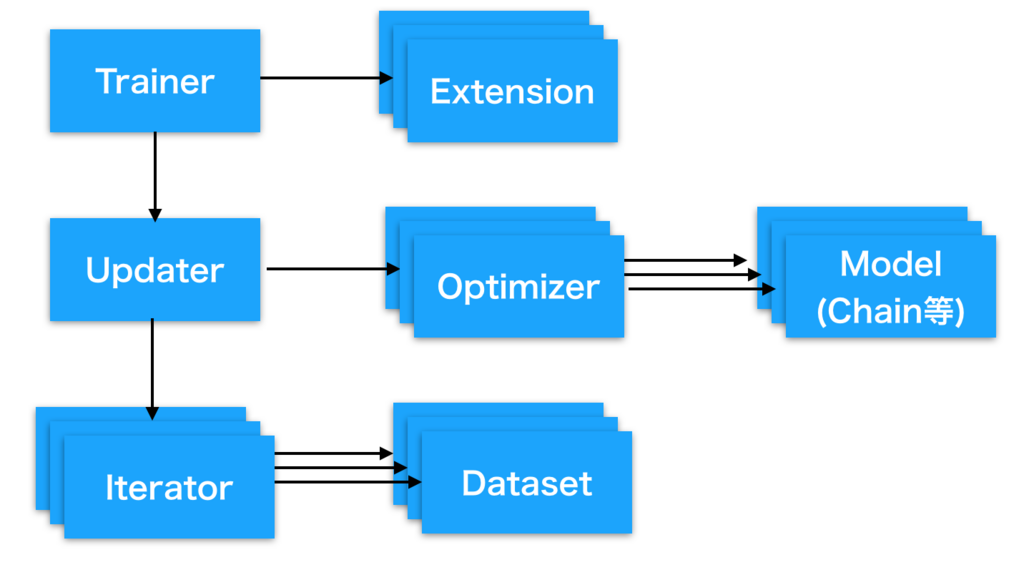
Trainer:
登録されたUpdaterを使って指定したepoch数やイテレーション数分だけ訓練を実行します。
Extensions:
Trainerが実行される過程で付属的に実行される処理を規定します。
モデルの評価を行うEvaluatorもExtensionの一種として定義されます。
Updater:
Datasetを含むIterator、訓練対象のモデルオブジェクトと勾配更新方法を規定するOptimizerを受け取って、 具体的にどのようにモデル群を更新していくかを規定します。
Optimizer:
SGD, Adamなど勾配計算後のパラメータ更新方法を規定します。
Model (Link / Chain)
ニューラルネットワーク(NN)の本体です。基本的にはLinkやFunctionを積み重ねてChainでラッピングしNNを構成していきます。 (Chain自体もLinkを継承して作られたLinkオブジェクトの一種なため、ChainとChainを繋いだりChainとLinkを繋いだりもできます)
Datasetと合わせてLinkやChainで作られるChainerのNNの概要を図にまとめてみました。

Iterator:
下記のデータセットを受け取り、ミニバッチサイズや取り出し順のポリシーを設定します。
Dataset:
画像などのデータとラベルの対を多数保持します。下記のエントリーでまとめたように、 Datasetクラスを継承することで極めて簡単かつ柔軟に定義できます
chainer: Evaluatorを自作してトレーニング中のモデルの評価を柔軟に行う
Evaluatorとは
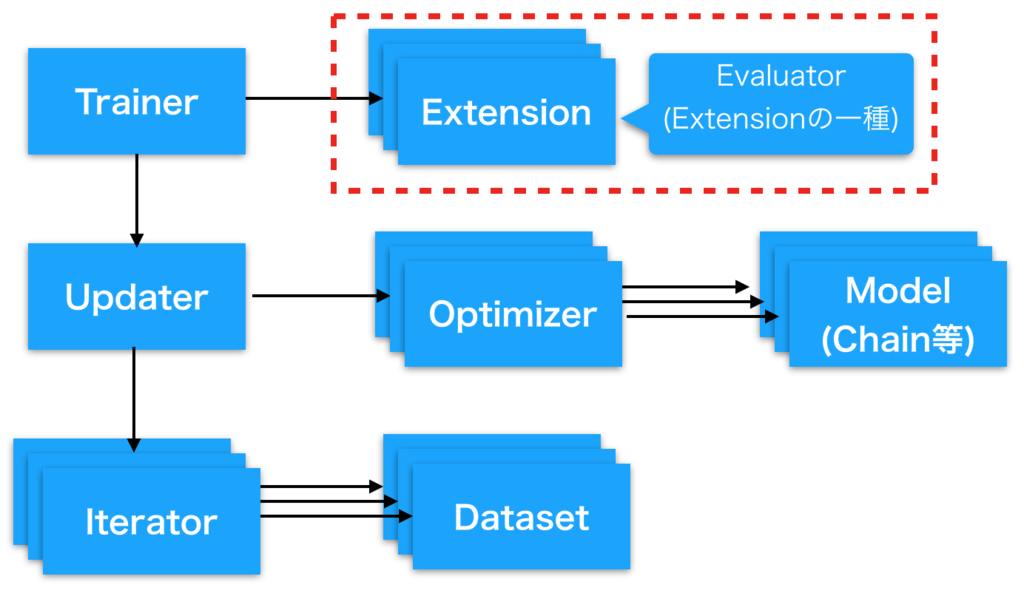
DNNの訓練を行う中でモデルの訓練が意図通り進んでいるかを評価したくなることが多いと思います。
Chainerでは定義したモデルの訓練を行う際にそのモデルの評価を行うための仕組みとしてEvaluatorという 仕組みを持っています。
このEvaluatorは 以前解説したExtensionの一種として作られています。
本質的にはExtensionを自分で自作すればモデルの評価ももちろん可能なのですが、 Evaluatorを継承してカスタマイズすることでaccuracyやlossの計上、イテレータの状態管理などをスマートに行うことができます。
chainer 標準のEvaluator
まず、継承元になるEvaluatorの作りを確認してみましょう。 Evaluatorについては、chainer標準のextensions.Evaluatorがかなり汎用的に作られており、自作せずに済むのならそれに越したことはないので。
Trainer extensions — Chainer 2.0.0 documentation
Evaluatorは、まず下記のようなオブジェクトを受け取ります。
- iterator: 評価用のデータセット、ミニバッチサイズ等が設定されたイテレータオブジェクト
- target: 評価対象となるモデル、もしくはモデルの列挙されたdict。
- converter: イテレータから取り出した(データ, ラベル)のタプルを訓練用のミニバッチに変換する関数
- device: 評価計算を行うために利用するGPU番号
- eval_hook: 評価前に実行される関数(なくてもok)
- eval_func: 評価を行うために呼び出される関数 (指定されない場合、targetに渡したモデルのcallが代わりに利用される)
chainer.training.extensions.evaluator — Chainer 2.0.0 documentation
で、それぞれ主要なメソッドの動作をざっくり確認すると
- __init__ :
- 渡された引数をインスタンス変数として格納する。
- 特に、targetにモデルのdictではなく単一のLinkが渡された場合、そのlinkを"main"という名前で辞書登録しなおす
- __call__:
- Reporter objectを作成して、targetとして渡された各リンクのを監視対象に指定する。
- evaluateメソッドを呼び出し、その結果をreporterを使ってreportする。 (__call__の戻り値は参照されておらず、reporter_module.reportに渡したdictが印字対象な点に注意)
- evaluate:
ということをしています。
つまり、標準のEvaluatorではこういうことが可能です。
- 単一のモデルと単一イテレータの評価。
- モデルのcallを評価対象にしても良いし、eval_funcで関数を渡して評価に使っても良い
標準のEvaluatorは複数のモデルをtargetに辞書として受け取ることはできますが、 evaluateメソッドが'main'のみを用いて評価するようになっているため、複数のモデルを評価に用いることはできません。
Evaluatorを自作する
逆に標準のEvaluatorではできないこと、例えば
- 複数のモデルやイテレータを使った評価(たとえばGANのGeneratorとDiscriminatorなど)
- accuracyやloss以外の指標値の出力(一応、eval_funcを使えば可能ですが)
などがしたい場合にはEvaluatorを自作すると良いかと思います。 Updaterの自作をした場合には対応するEvaluatorを作りたいことが多いかと思います。
拡張例
様々な拡張方法があると思いますので、やりたいことベースでchainer標準のEvaluatorを継承して独自のEvaluatorを定義する幾つか例を挙げていきます。
①とにかく指定した値をログやレポートに表示させたい
印字させたい項目を項目名をkey、スカラー値をvalueに持つ辞書をreporter_module.reportに渡せば、 とりあえず指定した値をログやレポートに表示させられます。
from chainer import reporter as reporter_module from chainer.training import extensions class MyEvaluator(extensions.Evaluator): def __call__(self, trainer=None): result = {"hoge": 4, "piyo": 88} reporter_module.report(result) return None
出力:
{ (略) "hoge": 4.0, "piyo": 88.0 }
chainerのReportで受け取る辞書(dict)の各値はスカラー値であることが必須です(文字列やリストは渡せません)。
②複数のモデルを用いて評価を行う
影響しあう複数のモデルを並列に訓練しているなどで評価を行いたい場合、 evaluateでtargetに指定するモデルをself._targetsから取り出す際に指定するorループで順に呼び出すなどすると良いと思います。
呼び出し元:
trainer.extend(MyEvaluator(test_iter, {"model1": model, "model2":model2}, device=args.gpu))
Evaluator側:
class MyEvaluator(extensions.Evaluator): default_name="myval" def evaluate(self): #target = self._targets['main'] summary = reporter_module.DictSummary() for name, target in six.iteritems(self._targets): iterator = self._iterators['main'] #target = self._targets['main'] eval_func = self.eval_func or target if self.eval_hook: self.eval_hook(self) if hasattr(iterator, 'reset'): iterator.reset() it = iterator else: it = copy.copy(iterator) #summary = reporter_module.DictSummary() for batch in it: observation = {} with reporter_module.report_scope(observation): in_arrays = self.converter(batch, self.device) with function.no_backprop_mode(): if isinstance(in_arrays, tuple): eval_func(*in_arrays) elif isinstance(in_arrays, dict): eval_func(**in_arrays) else: eval_func(in_arrays) summary.add(observation) return summary.compute_mean()
色々書いてあるように見えますが、元のevaluate()からの変更点は
- クラス変数default_nameを指定している(下記のログ出力のようにReporterがログ項目の接頭辞にしてくれます)
- target = self.targets['main']ではなくself.targetsからループで取り出すようにしている
- summaryの宣言をそのループの外側に書いた
だけです。
self._targetにはEvaluator定義時に指定したモデルが入っているのですが、
- 単一のモデル渡す(そのモデルが"main"という名前で__init__内で辞書登録される)
- モデルを辞書で渡す
のどちらでも良いようになっています。
このようにすることで
{ (略) "myval/model1/loss": 0.07286249771073926, "myval/model1/accuracy": 0.9748000055551529, "myval/model2/accuracy": 0.0888000001013279, "myval/model2/loss": 2.3258586740493774 }
のように各モデルに対する評価を出力できます。
③モデルの評価中に独自指標値を出力
Inceptionのように一つのモデルから複数の出力がある場合など、accuracyとloss以外の指標を計測してログに出力したいことも多いと思います。そのような場合には
def evaluate(self): iterator = self._iterators['main'] target = self._targets['main'] eval_func = self.eval_func or target if self.eval_hook: self.eval_hook(self) if hasattr(iterator, 'reset'): iterator.reset() it = iterator else: it = copy.copy(iterator) summary = reporter_module.DictSummary() for batch in it: observation = {} with reporter_module.report_scope(observation): in_arrays = self.converter(batch, self.device) with function.no_backprop_mode(): if isinstance(in_arrays, tuple): eval_func(*in_arrays) elif isinstance(in_arrays, dict): eval_func(**in_arrays) else: eval_func(in_arrays) summary.add({MyEvaluator.default_name + '/currenttime': int(time.time())}) print(observation) summary.add(observation) return summary.compute_mean()
summary.add({MyEvaluator.default_name + '/currenttime': int(time.time())})を足しています
これはreporter_module.report_scope(observation)のスコープ内でchainer.reporter.report(dict)が呼び出されると、
observationにdictが追加されるという仕組みを用いています
例ではUnixtimeをログに出していますが、モデルや出力に関する適切な数値を渡すことで 評価中のモデルについて都合の良い指標を出力できます。
(私の場合だと例えば、文字列を認識するモデルに対して正解文字列までの編集距離を出力するのに使っていました)
④GANのUpdater / Evaluator
下記の記事がGAN用のUpdater / Evaluatorの対の実装例になっているので、GANを実装したい方は参考に できると思います。
chainerで少し複雑なモデルを初めて扱うことになると評価をどうしようか迷うと思いますが(実際迷いました)、このようにEvaluatorを拡張することで柔軟に対処できるようになるかと思います。
chainer: Extensionを自作してディープラーニングの訓練に独自処理を挟み込む
なぜExtensionを自作するのか
Chainerのモデルのトレーニング中に、
- たまにモデルの出力をダンプさせたい(特に生成系で必要になる)
- 1エポック毎に学習率を手動で変更したい
など、独自の処理を定期的に挟みたくなることがあるかと思います。 このような願いを叶えるのがchainerのextensionという仕組みです。 extensionはtrainerに仕掛けておくことで指定した間隔ごとに独自に定義した処理を実行してくれます。
extensionはtrainerにひも付き、一つのtrainerにいくつでもextensionを設定することができます。
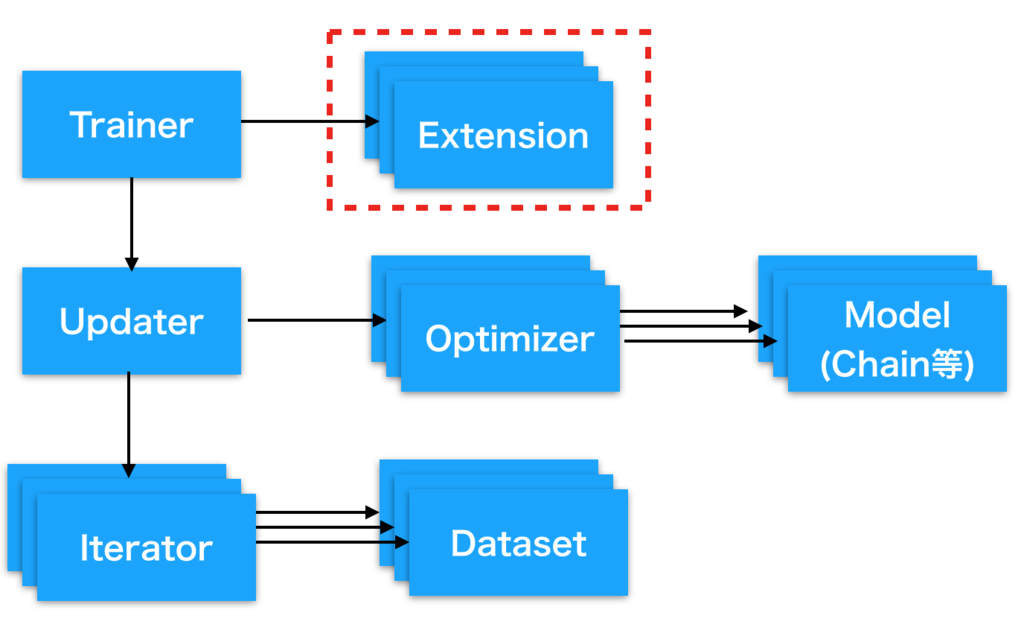
Extensionの自作方法
モデルを定期的に評価するEvaluatorもこのextensionを使って実装されています。 モデルの評価用途にはこのEvaluatorを拡張したほうが早いでしょう。 ここでは評価以外のより一般的にextensionを自作する方法にフォーカスします
例えば、公式ではこのようなExtensionが用意されています。よく使うのはこのあたりでしょうか。
| Extension名 | 概要 |
|---|---|
| extensions.Evaluator | モデルの損失や正解率を評価する |
| extensions.snapshot | モデルの重みをファイルに保存する |
| extensions.LogReport | Evaluator等で測定した各種の評価値をログに出力する |
| extensions.PrintReport | 指定した評価値を標準出力やファイルに出力する |
これらの公式Extensionはチュートリアル等でも言及されていますが、 extensionは簡単に自作することも可能です。
簡単なextensionの例として、「"hoge"とpirntするだけ」のextensionを作ってみましょう。
main.py | + lib -- print_hoge.py
このようなディレクトリ構成で、 定義側(print_hoge.py):
print_hoge.py def print_hoge(): @training.make_extension(trigger=(1, 'epoch')) def _print_hoge(trainer): print("hoge") return _print_hoge
呼び出し側 (main.py):
from lib.print_hoge import * (略) trainer = training.Trainer(updater, (30, 'epoch'), out=args.output) trainer.extend(print_hoge())
たったこれだけの記述で、1epochに一回、hogeと標準出力に出力されるようになります。
Extensionのカスタマイズ
動作する間隔を変えたければ
- trigger=(1, 'iteration'): 1イテレーションに1回
- trigger=(3, 'epoch'): 3エポックに一回
などtriggerを変更すればokです。
また、extensionの中でモデルを参照したければtrainer.update.modelとういうように、
trainerに登録されたupdater経由でアクセス可能です。
下記のように実行時に引数を与えることも可能です。
print_hoge.py def print_hoge(message): @training.make_extension(trigger=(1, 'epoch')) def _print_hoge(trainer): print(message) return _print_hoge 呼び出し側 (main.py) from lib.print_hoge import * (略) trainer = training.Trainer(updater, (30, 'epoch'), out=args.output) trainer.extend(print_hoge("fuga"))
後はextension名と中身の処理を好きなように書き換えればokです。
以下はもう少し詳しく知りたい方向け
書き方のポイントは、関数print_hoge()が関数「_print_hoge」自体を返り値に返却していることです。
実際、「関数を返却する関数」を定義しなくても
print_hoge.py @training.make_extension(trigger=(1, 'epoch')) def print_hoge(trainer): print("hoge")
と定義して、呼び出し側(main.py)で
from lib.print_hoge import * (略) trainer = training.Trainer(updater, (30, 'epoch'), out=args.output) trainer.extend(print_hoge)
とtrainer.extendに関数を直接渡せば最初の例と同じように動作します。
(trainer.extend(print_hoge())ではなくtrainer.extend(print_hoge)となっていることに注意してください)
上記のようにも書けるのですが、chainerの公式コードでは一度「関数を返す関数(ないしクラス)」を使う実装がなされています。
これは外側の関数(print_hoge)を関数と内側の関数(_print_hoge)の間で変数を定義することで_print_hogeで参照する変数のクロージャを作ることができ、色々便利だからかと思います。
また、
@training.make_extension(trigger=(1, 'epoch')) def print_hoge(trainer): print("hoge") @training.make_extension(trigger=(1, 'epoch'))
という処理も一見何か難しいことをしているように見えますが、
make_extension関数内で行われていることは下記とほぼ同義です。
def sample_recog(trainer): print("hoge") sample_recog.trigger=(1, 'iteration')
つまり、極端な話
def sample_recog(): def _sample_recog(trainer): print("hoge") _sample_recog.trigger=(1, 'iteration') return _sample_recog
このように定義してもmake_extensionを使ったときと同じように動きます。